شبکه تنظیمکننده ژن یا شبکه تنظیمکننده ژنتیک (GRN)
1. مقدمه
-
تعریف شبکههای تنظیم ژن (Gene Regulatory Networks – GRNs)
-
تاریخچه توسعه مفاهیم GRN
-
اهمیت این شبکهها در زیستشناسی سامانهای (Systems Biology)
2. مبانی زیستی
-
DNA، RNA، پروتئینها و نقش آنها در تنظیم
-
عناصر تنظیمی سیس (Cis-regulatory elements): شامل پروموترها، تقویتکنندهها (Enhancers)، خاموشکنندهها (Silencers)، و ایزولاتورها (Insulators)
-
عوامل تنظیمی ترانس (Trans-regulatory factors): شامل فاکتورهای رونویسی، همفاکتورها، و پروتئینهای متصلشونده به RNA
-
تنظیمات اپیژنتیک (Epigenetic regulation): شامل متیلاسیون DNA، تغییرات هیستونی، و بازآرایی کروماتین
-
تنظیم پس از رونویسی (Post-transcriptional regulation): شامل برش و اتصال RNA (Splicing)، میکرو RNAها (miRNAs)، و پایداری RNA
-
تنظیم پس از ترجمه (Post-translational regulation)
3. معماری شبکه
-
گرهها (Nodes) و یالها (Edges) در GRNها
-
سازماندهی سلسلهمراتبی و ماژولار
-
حلقههای پیشخور (Feedforward) و بازخور (Feedback)
-
سوئیچها و نوسانسازها (Oscillators)
-
الگوهای تکرارشونده شبکه (Network motifs) و اهمیت عملکردی آنها
-
نمونهها در سیستمهای پروکاریوتی و یوکاریوتی
4. پویاییهای GRN
-
تنظیم وابسته به زمان
-
مسیرهای انتقال سیگنال و ارتباط آنها با GRNها
-
نشانههای محیطی و رشدی (Environmental & Developmental Cues)
-
نویز در بیان ژن و مدلسازی تصادفی (Stochastic modeling)
5. رویکردهای تجربی برای نقشهبرداری GRN
-
ایمونواِیماونوپریسیپیتاسیون کروماتین (ChIP-seq, ChIP-exo)
-
DNAse-seq و ATAC-seq برای نقشهبرداری کروماتین باز
-
RNA-seq و تکسلولی RNA-seq
-
دستکاریهای مبتنی بر CRISPR/Cas9
-
پروتئومیکس و آزمونهای برهمکنش پروتئین–DNA (مثل Yeast One-Hybrid)
-
Perturb-seq و آزمونهای گزارشگر با توان بالا (Massively Parallel Reporter Assays)
6. مدلسازی محاسباتی GRN
-
ادغام دادهها از منابع مختلف اُمیکس (Omics)
-
الگوریتمهای استنتاج شبکه: مبتنی بر همبستگی، رگرسیون، نظریه اطلاعات، شبکههای بیزی، مدلهای معادلات دیفرانسیل معمولی (ODE-based)، و شبکههای بولی (Boolean networks)
-
یادگیری ماشین و روشهای یادگیری عمیق (Deep Learning)
-
مدلسازی پویا (Dynamic) در مقابل استنتاج ایستا (Static)
-
تخمین پارامترها و اعتبارسنجی مدل
-
پلتفرمها و نرمافزارهای شبیهسازی
7. GRN در رشد و تمایز سلولی
-
رشد جنینی (Embryonic Development)
-
پلوریپوتنسی سلولهای بنیادی و مشخص شدن سرنوشت سلولی (Lineage Specification)
-
بازآرایی GRN در فرآیند تمایز
-
ارگانیسمهای مدل: C. elegans، مگس سرکه (Drosophila)، زِبرا فیش (Zebrafish)، و موش
8. GRN در سلامت و بیماری
-
شبکههای تنظیمی در سرطان
-
GRN در پاسخ ایمنی
-
شبکههای بیماریهای عصبیتخریبی (Neurodegenerative)
-
شبکههای برهمکنش پاتوژن–میزبان
9. زیستفناوری ترکیبی و مهندسی GRN
-
طراحی مدارهای ژنی مصنوعی
-
کاربردها در زیستفناوری و درمان
-
مطالعات موردی: سوئیچهای دوحالته (Toggle Switches)، نوسانسازها (Oscillators)، دروازههای منطقی (Logic Gates)
-
ملاحظات ایمنی و اخلاقی
10. GRN در بستر تکامل
-
تکامل و بازآرایی شبکهها
-
ژنومیکس مقایسهای عناصر تنظیمی
-
پایداری (Robustness) و قابلیت تکامل (Evolvability) شبکهها
11. چالشها، محدودیتها و مسیرهای آینده
-
محدودیتهای تجربی
-
یکپارچهسازی و استانداردسازی دادهها
-
قابلیت مقیاسپذیری روشهای محاسباتی
-
چشمانداز برای نقشهبرداری GRN در سطح تکسلول، فضایی و زمانی
-
فناوریهای نوظهور
12. منابع و مطالعات بیشتر
-
کتابشناسی جامع
-
مقالات مروری کلیدی و پژوهشهای پیشگام
1. مقدمه
شبکههای تنظیم ژن (Gene Regulatory Networks – GRNs) یکی از بنیادیترین چارچوبهای مفهومی در زیستشناسی مولکولی مدرن و زیستشناسی سامانهای (Systems Biology) به شمار میروند. در اصل، GRNها توصیفکنندهی شبکههای پیچیده تعاملات مولکولی هستند که فعالیت رونویسی ژنها را کنترل میکنند و با ادغام سیگنالهای محیط درونسلولی و محرکهای خارجی، عملکردهای سلول را هماهنگ میسازند.
یک GRN را میتوان به عنوان یک شبکه پیچیده تصور کرد که در آن گرهها (Nodes) نمایانگر ژنها، فاکتورهای رونویسی یا سایر مولکولهای تنظیمی هستند و یالها (Edges) نشاندهنده روابط تنظیمی میان آنها. این روابط معمولاً جهتدار هستند، زیرا یک مولفه (مثل فاکتور رونویسی) میتواند بیان ژن دیگری را فعال یا سرکوب کند. برخلاف یک فهرست ساده از جفتهای ژن–پروتئین، GRNها منطق پویا، بههمپیوسته و اغلب غیرخطی موجودات زنده را در حفظ همایستایی (Homeostasis)، پاسخ به تغییرات محیطی، و اجرای برنامههای پیچیده تکوینی نشان میدهند.
تحول مطالعه GRNها
مطالعه GRNها در چند دهه اخیر دگرگونی شگرفی پیدا کرده است. زیستشناسی مولکولی اولیه دیدگاهی ژنبهژن ارائه میداد که عمدتاً از طریق آزمایشهای بیوشیمیایی و ژنتیکی پرزحمت و متمرکز بر مسیرهای منفرد حاصل میشد. این مطالعات پایهای بودند — مانند کار کلاسیک ژاکوب و مونو (Jacob & Monod) روی اپرون لاک (lac operon) در اشریشیا کلی (E. coli) — اما تنها تصویری محدود از پیچیدگی تنظیم سلولی ارائه میکردند.
با آغاز ژنومیک و ترنسکریپتومیکس با توان بالا در اوایل قرن بیستویکم، این حوزه دگرگون شد. فناوریهایی مانند ریزآرایههای DNA (DNA microarrays) و بعدها توالییابی RNA (RNA-seq) و ایمونواِیماونوپریسیپیتاسیون کروماتین همراه با توالییابی (ChIP-seq)، امکان ثبت تعاملات تنظیمی را در مقیاس کل ژنوم فراهم کردند. اخیراً، ترنسکریپتومیکس تکسلولی و ترنسکریپتومیکس فضایی لایههای جدیدی از وضوح را اضافه کردهاند که اجازه بررسی برنامههای تنظیمی در سطح تکسلول و جایگاه مکانی آنها در بافت را میدهد.
اهمیت زیستی GRNها
اهمیت زیستی GRNها غیرقابل اغراق است. هر فرایند سلولی — از تنظیم متابولیک تا پیشرفت چرخه سلولی، از پاسخهای ایمنی تا سیگنالدهی عصبی — تحت تاثیر کنش هماهنگ شبکههای تنظیمی است.
-
در تکوین جنینی، GRNها فعالسازی و سرکوب متوالی مجموعه ژنها را هدایت میکنند تا سلولها را از طریق تعیین سرنوشت دقیق، حرکات مورفوژنتیکی و تمایز بافتی پیش ببرند.
-
در سلولهای ایمنی، GRNها پاسخهای رونویسی سریع را در برابر پاتوژنها سازماندهی میکنند و همزمان تحمل نسبت به آنتیژنهای خودی را حفظ میکنند.
-
در سیستم عصبی، آنها پایهگذار فرآیندهایی مانند انعطافپذیری سیناپسی (Synaptic Plasticity) و شکلگیری حافظه هستند.
در همه این موارد، ساختار شبکهای موجب مقاومت در برابر اختلالات، تضمین زمانبندی مناسب و امکان ادغام چندین سیگنال میشود.
سازمان ماژولار و سلسلهمراتبی
یکی از ویژگیهای مهم GRNها سازمان ماژولار و سلسلهمراتبی آنهاست.
-
ماژولها گروهی از ژنها و تنظیمکنندهها هستند که برای انجام یک عملکرد خاص با هم کار میکنند و اغلب در زمینههای زیستی مختلف حفظ شدهاند.
-
سلسلهمراتب زمانی ایجاد میشود که تنظیمکنندههای اصلی (Master Regulators) مجموعه وسیعی از ژنهای پاییندستی را کنترل میکنند.
این ساختار مزایای تکاملی و عملکردی دارد:
-
امکان استفاده مجدد از ماژولهای تنظیمی در زمینههای مختلف
-
سازگاری سریع با تغییرات محیطی
-
کاهش تداخلهای زیانآور بین مسیرهای نامرتبط
الگوهای تکرارشونده شبکهای (Network Motifs) مانند حلقههای پیشخور (Feedforward)، بازخور منفی (Negative Feedback) و سوئیچهای دوحالته (Bistable Switches) دارای ویژگیهای دینامیکی خاص هستند.
مثلاً:
-
حلقههای پیشخور میتوانند نویز سیگنالهای بالادستی را فیلتر کنند.
-
سوئیچهای دوحالته به سلولها امکان میدهند حالتهای پایدار و متمایز را اتخاذ کنند.
دیدگاه زیستشناسی سامانهای
از دیدگاه زیستشناسی سامانهای، GRNها تنها نقشههای توصیفی نیستند، بلکه مدلهای پیشبینیکننده رفتار سلول هستند. این توان پیشبینی ناشی از آن است که توپولوژی شبکه و منطق تنظیمی را میتوان ریاضیسازی کرد تا واکنش سلول به تغییرات محیطی، جهشهای ژنتیکی یا مداخلات دارویی شبیهسازی شود.
این مدلها در حوزههایی مثل:
-
زیستفناوری ترکیبی (Synthetic Biology) برای طراحی مدارهای ژنی مصنوعی
-
پزشکی شخصی برای مدلسازی اختصاصی GRN بیمار
ارزش بالایی دارند.
چالشهای بازسازی دقیق GRNها
بازسازی دقیق GRNها همچنان یک چالش بزرگ است، زیرا:
-
سیستمهای زیستی ذاتاً پر از نویز هستند.
-
تنظیم رونویسی علاوه بر توالی DNA، تحت تأثیر عوامل زیادی مثل وضعیت کروماتین، پایداری RNA، تغییرات پس از ترجمه و فعالیت RNAهای غیرکدکننده است.
-
تعاملات تنظیمی وابسته به زمینه هستند؛ مثلاً یک فاکتور رونویسی در یک نوع سلول فعالکننده و در نوع دیگر سرکوبگر است.
به همین دلیل، دادههای تجربی تنها تصاویر لحظهای از یک واقعیت بسیار پویا و سیال ارائه میدهند. برای حل این مشکل، محققان به طور فزاینده لایههای مختلف دادههای اُمیکس — ترنسکریپتومیکس، اپیژنومیکس، پروتئومیکس و متابولومیکس — را ادغام میکنند تا هم جنبههای ایستا و هم جنبههای پویای تنظیم را ثبت کنند.
پیوند GRN با بیماریها
بسیاری از بیماریها را میتوان به عنوان اختلال در شبکههای تنظیمی درک کرد:
-
در سرطان، جهشها در فاکتورهای رونویسی، بازآرایندههای کروماتین یا مسیرهای سیگنالدهی میتوانند GRNها را بازسیمکشی کنند و منجر به تکثیر کنترلنشده سلول، فرار از آپوپتوز و متاستاز شوند.
-
در بیماریهای عصبیتخریبی، تغییر در برنامههای تنظیمی ممکن است باعث اختلال در نگهداری نورونها یا عملکرد سیناپسی شود.
-
حتی در بیماریهای عفونی، پاتوژنها اغلب شبکههای تنظیمی میزبان را ربوده و به نفع خود تغییر میدهند.
شناسایی گرههای کلیدی در GRNهای مرتبط با بیماری، مسیرهای جدیدی برای هدفگیری درمانی فراهم میکند.
دیدگاه تکاملی
از منظر تکامل، GRNها ابزاری قدرتمند برای درک چگونگی ایجاد صفات و فنوتیپهای پیچیده هستند. تغییرات در عناصر تنظیمی سیس (Cis-regulatory elements) — نه لزوماً در توالیهای کدکننده — نقش مهمی در تنوع مورفولوژیک گونهها دارند.
ماهیت ماژولار GRNها آنها را برای تغییرات تکاملی مناسب میسازد، زیرا ماژولها میتوانند در زمینههای جدید به کار گرفته شوند بدون اینکه سایر عملکردها مختل شوند.
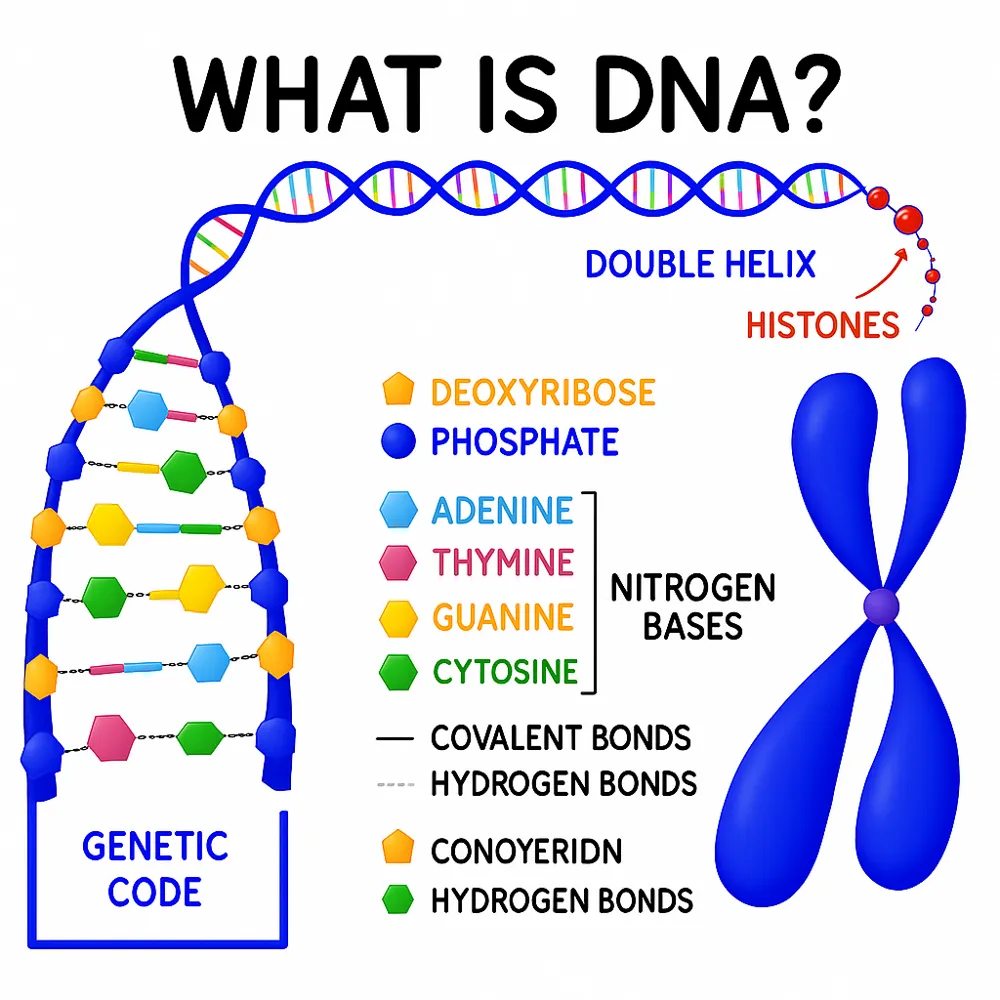
۲. مبانی زیستی شبکههای تنظیم ژن (GRNs)
درک شبکههای تنظیم ژن (GRNs) نیازمند شناخت عمیق از اجزای مولکولی بنیادی و سازوکارهایی است که در پسِ بیان ژن قرار دارند. GRNها در اصل مدلهای انتزاعی از تعاملات مولکولی واقعی هستند و دقت این مدلها بستگی دارد به این که تا چه حد فرآیندهای واقعی زیستی داخل سلول را به درستی میشناسیم.
این بخش به بررسی بلوکهای سازنده اصلی GRNها میپردازد؛ از دگم مرکزی زیستشناسی مولکولی آغاز کرده و سپس به لایههای متعدد تنظیمی میرسد که به طور مشترک الگوهای مکانی–زمانی فعالیت ژنها را تعیین میکنند.
۲.۱ دگم مرکزی و پیامدهای تنظیمی آن
دگم مرکزی زیستشناسی مولکولی که نخستین بار توسط فرانسیس کریک در سال ۱۹۵۸ مطرح شد، بیان میکند که اطلاعات ژنتیکی به صورت جهتدار از DNA → RNA → پروتئین جریان مییابد. این مفهوم اگرچه ساده به نظر میرسد، اما واقعیت این است که کنترل تنظیمی در هر مرحله بسیار پیچیده است.
در یک GRN، هر مرحله — از آغاز رونویسی تا تغییرات پس از ترجمه — یک نقطه بالقوه برای تنظیم است، و به سلول این امکان را میدهد که بیان ژنها را دقیقاً بر اساس نیازهای داخلی یا محرکهای خارجی تنظیم کند.
-
تنظیم رونویسی: فاکتورهای رونویسی (TFs) به توالیهای خاص DNA در پروموترها یا تقویتکنندهها (Enhancers) متصل میشوند تا اتصال RNA پلیمراز را تقویت یا مهار کنند. این اتصال اغلب همکارانه است، شامل چندین TF و همفاکتور، و به وضعیت کروماتین حساس است.
-
پس از رونویسی، مولکولهای RNA دچار اسپلایسینگ (Splicing)، کپگذاری (Capping) و پلیآدنیلاسیون (Polyadenylation) میشوند که هر کدام میتوانند تنظیم شوند تا از یک ژن، ایزوفرمهای مختلف RNA تولید شود.
-
ترجمه نیز تحت کنترل مکانیسمهایی مثل چارچوبهای بازخوانی بالادست (uORFs)، ساختار ثانویه RNA و دسترسپذیری فاکتورهای آغاز ترجمه است.
-
در نهایت، پروتئینها میتوانند پس از ترجمه دچار تغییراتی مانند فسفریلاسیون، یوبیکویتیناسیون، متیلاسیون و سایر اصلاحات شوند که پایداری، مکان یا فعالیت آنها را تغییر میدهد.
۲.۲ عناصر تنظیمی سیس (Cis-regulatory elements): پروموترها، تقویتکنندهها، خاموشکنندهها و ایزولاتورها
عناصر تنظیمی سیس توالیهای غیرکدکننده DNA هستند که رونویسی ژنهای نزدیک به خود را کنترل میکنند. واژه سیس به این معناست که این عناصر روی همان مولکول DNA که در آن قرار دارند، اثر میگذارند.
-
پروموترها (Promoters): محل اصلی آغاز رونویسی هستند و درست در بالادست نقطه شروع رونویسی (TSS) قرار دارند. آنها شامل موتیفهای اصلی مثل جعبه TATA، توالیهای Inr و عناصر پروموتر پاییندستی (DPE) هستند که توسط ماشین رونویسی پایه شناسایی میشوند. فعالیت پروموتر به وسیله عناصر پروموتر مجاور و عناصر تنظیمی دورتر تعدیل میشود.
-
تقویتکنندهها (Enhancers): توالیهای سیس دوردستی هستند که میتوانند بهطور چشمگیری نرخ رونویسی را افزایش دهند، معمولاً به شکل اختصاصی برای نوع سلول یا مرحله تکوینی. آنها مستقل از جهتگیری عمل میکنند و میتوانند حتی صدها کیلوباز دورتر از ژن هدف باشند، اما از طریق حلقهزایی کروماتین به نزدیکی آن آورده میشوند.
-
خاموشکنندهها (Silencers): عملکردی مشابه تقویتکنندهها دارند ولی رونویسی را سرکوب میکنند. آنها پروتئینهای سرکوبگر و همسرکوبگر را جذب میکنند که باعث تراکم کروماتین یا اختلال در مونتاژ ماشین رونویسی میشود. خاموشکنندهها برای حفظ هویت سلولی اهمیت دارند چون از بیان ژنهای مسیرهای جایگزین جلوگیری میکنند.
-
ایزولاتورها (Insulators): عناصر سیس تخصصی هستند که به عنوان مرزهای ژنومی عمل میکنند. آنها میتوانند تعامل بین یک تقویتکننده و پروموتر را مسدود کنند یا از گسترش هتروکروماتین جلوگیری کنند. پروتئین CTCF یکی از شناختهشدهترین پروتئینهای متصلشونده به ایزولاتور در مهرهداران است و نقش مهمی در سازماندهی سهبعدی دامنههای کروماتین دارد.
۲.۳ عوامل تنظیمی ترانس (Trans-regulatory factors): فاکتورهای رونویسی، همفاکتورها و پروتئینهای متصلشونده به RNA
در حالی که عناصر سیس آدرسهای تنظیمی در ژنوم را مشخص میکنند، عوامل تنظیمی ترانس پروتئینها و RNAهایی هستند که این توالیها را شناسایی و تفسیر میکنند.
-
فاکتورهای رونویسی (TFs): شناختهشدهترین عوامل ترانس هستند. معمولاً شامل یک یا چند دامنه اتصال به DNA (DBD) برای شناسایی موتیفهای خاص و دامنههای فعالکننده یا سرکوبکننده برای تعامل با سایر پروتئینها هستند. خانوادههای TF بر اساس ساختار DBD تقسیمبندی میشوند؛ مثل پروتئینهای انگشت روی (Zinc Finger)، مارپیچ–گردش–مارپیچ (Helix-turn-helix)، بیسک لوسین زیپر (bZIP) و هومودومین (Homeodomain). بسیاری از TFها توسط مسیرهای سیگنالدهی تنظیم میشوند و این موضوع به GRNها امکان میدهد تا اطلاعات محیطی را یکپارچه کنند.
-
همفاکتورها (Cofactors): پروتئینهایی که به طور مستقیم به DNA متصل نمیشوند اما رونویسی را تعدیل میکنند. همفعالکنندهها مثل کمپلکس Mediator یا p300/CBP، RNA پلیمراز II را جذب کرده و هیستونها را اصلاح میکنند تا رونویسی تسهیل شود. همسرکوبگرها مثل NCoR یا کمپلکسهای دارای HDAC، علائم فعالکننده هیستون را حذف کرده یا علائم سرکوبگر اضافه میکنند.
-
پروتئینهای متصلشونده به RNA (RBPs): بیان ژن را در سطح پسارونویسی کنترل میکنند؛ از جمله اسپلایسینگ، انتقال، مکانیابی، پایداری و ترجمه RNA. آنها با اتصال به موتیفهای خاص در mRNA میتوانند بیان مجموعهای از ژنهای مرتبط عملکردی را هماهنگ کنند.
۲.۴ تنظیمات اپیژنتیک: تغییرات کروماتین و DNA
بیان ژن همچنین تحت تاثیر مکانیسمهای اپیژنتیک قرار دارد — تغییرات ارثپذیر در فعالیت ژن بدون تغییر در توالی DNA. این تنظیمات بیشتر از طریق تغییرات شیمیایی DNA و پروتئینهای هیستونی و همچنین تغییر در ساختار کروماتین انجام میشوند.
-
متیلاسیون DNA: معمولاً در کربن ۵ سیتوزین داخل دینوکلئوتیدهای CpG رخ میدهد و با سرکوب رونویسی در پروموترها مرتبط است. این تغییر میتواند مانع اتصال فعالکنندهها شود یا پروتئینهای متصلشونده به CpG متیله را جذب کند که کروماتین را متراکم میکنند.
-
تغییرات هیستونی: در اسیدهای آمینه خاص دمهای هیستونی رخ میدهد و شامل استیلاسیون، متیلاسیون، فسفریلاسیون، یوبیکویتیناسیون و سومویلاسیون است. به عنوان مثال:
-
H3K4me3 معمولاً با پروموترهای فعال مرتبط است.
-
H3K27me3 علامت سرکوب وابسته به کمپلکس Polycomb است.
فرضیه کد هیستونی (Histone Code) بیان میکند که ترکیب این علائم یک زبان تنظیمی پیچیده ایجاد میکند که توسط پروتئینهای مرتبط با کروماتین خوانده میشود.
-
-
کمپلکسهای بازآرایی کروماتین مثل خانوادههای SWI/SNF یا ISWI با هیدرولیز ATP، نوکلئوزومها را جابجا، حذف یا بازسازی میکنند تا دسترسپذیری DNA را تغییر دهند. این فرآیند برای اتصال فاکتورهای رونویسی به سایتهای هدف در کروماتین فشرده ضروری است.
۲.۵ تنظیم پساترنسکریپت (Post-Transcriptional Regulation)
وقتی یک ژن به یک رونوشت RNA اولیه (primary RNA transcript) تبدیل میشود، سرنوشت نهایی و نقش عملکردی آن در سلول توسط مجموعهای از مکانیسمهای پساترنسکریپت تعیین میشود. این فرآیندها یک لایه اضافی کنترل در شبکههای تنظیم ژن (GRNs) فراهم میکنند تا بیان ژن فراتر از مرحله آغاز رونویسی کنترل شود و سلول بتواند واکنشهای خود را با دقت مکانی و زمانی تنظیم کند.
یکی از مهمترین این مراحل، برش و وصله RNA (RNA splicing) است. در یوکاریوتها، RNA پیامرسان پیشساز (pre-mRNA) شامل اگزونها (کدکننده پروتئین) و اینترونها (غیرکدکننده) است. حذف اینترونها و اتصال اگزونها توسط اسپلایسوزوم (splicesome) انجام میشود که یک کمپلکس بزرگ ریبونوکلئوپروتئینی است.
اسپلایسینگ جایگزین (Alternative splicing) این امکان را فراهم میکند که چندین ایزوفرم پروتئینی از یک ژن تولید شود و ظرفیت عملکردی ژنوم افزایش یابد. این موضوع باعث میشود GRNها بتوانند خروجیهای خاص بافتی یا مرحلهای ایجاد کنند.
مثلاً یک ژن فاکتور رونویسی ممکن است ایزوفرمهایی تولید کند که تفاوت در دامنه اتصال به DNA یا نواحی تنظیمی دارند و در نتیجه در بافتها یا مراحل رشد مختلف، کنترل متفاوتی بر ژنهای هدف اعمال کنند.
ویرایش RNA (RNA editing) یکی دیگر از تغییرات پساترنسکریپت است که در آن نوکلئوتیدهای خاص در RNA پس از رونویسی تغییر میکنند.
-
ویرایش آدنوزین به اینوزین (A-to-I) که توسط آنزیمهای ADAR انجام میشود، میتواند توالی پروتئین را بازکدگذاری کند، الگوهای اسپلایسینگ را تغییر دهد یا پایداری RNA را دگرگون کند.
-
ویرایش سیتیدین به یوریدین (C-to-U) که کمتر رایج است، میتواند به همین ترتیب بر کدگذاری پروتئین و عناصر تنظیمی در RNA تأثیر بگذارد.
پایداری mRNA نقش مهمی دارد، زیرا نرخ تجزیه mRNA تعیین میکند که چه مدت یک مولکول RNA میتواند به پروتئین ترجمه شود. ناحیههای ۵′ و ۳′ UTR حاوی عناصر توالی خاص مثل عناصر غنی از AU (AREs) هستند که محل اتصال پروتئینهای متصلشونده به RNA (RBPs) یا میکروRNAها (miRNAs) میباشند و میتوانند باعث پایدار شدن یا تجزیه mRNA شوند. با تغییر انتخابی نیمهعمر mRNA، سلولها میتوانند سطح پروتئین را سریع تغییر دهند بدون اینکه نرخ رونویسی را دستکاری کنند.
مکانیابی mRNA (mRNA localization) تضمین میکند که RNAها به بخشهای خاصی از سلول منتقل شوند و ترجمه بهصورت محلی انجام شود. این موضوع در سلولهای بسیار قطبیشده مثل نورونها اهمیت ویژه دارد، جایی که mRNAها به دندریتها یا پایانههای آکسونی منتقل میشوند تا پروتئینسازی سریع در پاسخ به سیگنالهای سیناپسی انجام شود.
۲.۶ تنظیم در سطح ترجمه (Translational Regulation)
ترجمه که طی آن پروتئین از روی قالب mRNA ساخته میشود، یکی دیگر از نقاط کلیدی کنترل بیان ژن است. GRNها میتوانند ترجمه را از طریق مکانیسمهای مختلف کنترل کنند تا تولید پروتئین به سرعت و در پاسخ به تغییرات محیطی، استرس یا سیگنالهای خاص تغییر کند.
کنترل ترجمه سراسری معمولاً در پاسخ به استرس یا کمبود مواد مغذی اتفاق میافتد. مثلاً فسفریلاسیون فاکتور آغازگر یوکاریوتی ۲ آلفا (eIF2α) باعث کاهش آغاز ترجمه در پاسخ به استرس میشود تا منابع ذخیره گردد، در حالی که ترجمه mRNAهای خاص مرتبط با پاسخ به استرس همچنان ادامه یابد.
کنترل ترجمه اختصاصی هر RNA به عناصر موجود در خود mRNA وابسته است:
-
چارچوبهای بازخوانی آغازین (uORFs) در ناحیه ۵′ UTR میتوانند ترجمه توالی اصلی کدکننده را در شرایط عادی مهار کنند ولی در شرایط استرس، با تغییر مکانیسم آغاز ترجمه، اجازه بیان آن را بدهند.
-
ناحیههای ورودی داخلی ریبوزوم (IRESs) امکان آغاز ترجمه بهصورت مستقل از کلاهک (cap-independent) را فراهم میکنند، که در شرایطی که ترجمه وابسته به کلاهک متوقف شده است، اهمیت دارد.
میکروRNAها (miRNAs) که RNAهای کوچک و غیرکدکننده هستند، با اتصال به توالیهای مکمل در ناحیه ۳′ UTR هدف، باعث سرکوب ترجمه یا تجزیه mRNA میشوند. یک miRNA میتواند چندین mRNA را کنترل کند و هر mRNA نیز میتواند هدف چندین miRNA باشد، که این موضوع یک شبکه پیچیده تنظیم پساترنسکریپت ایجاد میکند.
۲.۷ تنظیم پساترنسلی (Post-Translational Regulation)
حتی بعد از ساخته شدن پروتئینها، عملکرد، پایداری و مکان آنها میتواند بهطور گسترده توسط تغییرات پساترنسلی (PTMs) تنظیم شود. این تغییرات به GRNها اجازه میدهند که فعالیت پروتئینهای تنظیمی، بهخصوص فاکتورهای رونویسی را در زمان واقعی تنظیم کنند.
-
فسفریلاسیون (Phosphorylation) که توسط کینازها انجام و توسط فسفاتازها حذف میشود، میتواند آنزیمها را فعال یا غیرفعال کند، تعاملات پروتئینی را تغییر دهد یا مکان پروتئین در سلول را جابهجا کند. مسیرهای انتقال سیگنال معمولاً با فسفریلاسیون فاکتورهای رونویسی پایان مییابند و فعالیت GRN را سریع تغییر میدهند.
-
یوبیکویتیناسیون (Ubiquitination) اتصال مولکولهای یوبیکویتین به لیزینهای پروتئین هدف است که اغلب آن را برای تجزیه توسط پروتئازوم علامتگذاری میکند. این مکانیسم امکان تجزیه سریع پروتئینهای تنظیمی را فراهم میکند و موجب تغییرات پویا در وضعیت شبکه میشود.
-
استیلاسیون (Acetylation) معمولاً در لیزین رخ میدهد و میتواند اتصال پروتئین به DNA یا پروتئینهای دیگر را تغییر دهد. مثلاً استیلاسیون هیستونها اغلب باعث بازتر شدن کروماتین و تسهیل رونویسی میشود.
سایر تغییرات مانند متیلاسیون، SUMOylation و لیپیداسیون نیز لایههای دیگری از تنظیم را اضافه میکنند و یک پروتئین میتواند همزمان چندین حالت تنظیمی داشته باشد.
۲.۸ ادغام لایههای مختلف تنظیم
در عمل، GRNها بهصورت جداگانه در یک سطح تنظیمی عمل نمیکنند؛ بلکه ترکیبی از کنترلهای رونویسی، پساترنسکریپت، ترجمهای و پساترنسلی را در یک سیستم چندلایه و هماهنگ ادغام میکنند.
مثلاً در پاسخ ایمنی، فعالسازی رونویسی ژنهای سیتوکین به سرعت با اسپلایسینگ جایگزین، تنظیم پایداری mRNA و فسفریلاسیون واسطههای سیگنالدهنده همراه میشود. یا در توسعه جنینی، فاکتورهای رونویسی آبشاری از فعالسازی ژنها را شروع میکنند، در حالی که miRNAها جزئیات الگوهای بیان را دقیق میکنند و PTMها فعالیت پروتئینها را با مرحله رشد هماهنگ میسازند.
۲.۹ تنظیم مکانی و وابسته به بافت یا شرایط
GRNها به شدت وابسته به زمینه (Context-dependent) هستند، به این معنا که یک عامل تنظیمی ممکن است بسته به نوع سلول، مرحله رشد یا شرایط محیطی، اثرات متفاوتی داشته باشد. وضعیت کروماتین، حضور یا عدم حضور کوفاکتورها و ورودیهای سیگنالدهی همه بر عملکرد یک فاکتور رونویسی یا پروتئین متصل به RNA اثر میگذارند.
تنظیم مکانی نیز بسیار مهم است. درون یک بافت، سلولها میتوانند در ریزمحیطهای متفاوتی قرار گیرند که آنها را در معرض سیگنالها یا محرکهای مکانیکی متفاوت قرار میدهد و باعث تفاوتهای موضعی در فعالیت GRN میشود. در سازماندهی بافتها و اندامها، شیبهای مورفوژن میتوانند الگوهای مکانی بیان ژن را ایجاد کنند.
این ویژگی اختصاصی بودن مکانی و زمینهای باعث میشود GRNها بتوانند در محیطهای بیولوژیکی مختلف دوباره استفاده شوند بدون آنکه نتایج یکسانی تولید کنند، که این اصل برای توسعه و تکامل بسیار بنیادی است.
۳. معماری شبکه
شبکههای تنظیم ژن (Gene Regulatory Networks - GRNs) را میتوان نهتنها بر اساس اجزای منفردی که عملکردهای مولکولی مشخصی انجام میدهند، بلکه از نظر سازماندهی توپولوژیکی کلی آنها نیز درک کرد — یعنی نحوه چیدمان گرهها (ژنها، فاکتورهای رونویسی، RNAهای تنظیمی، مولکولهای سیگنالدهنده) و یالها (تعاملات تنظیمی) که آنها را به هم متصل میکنند.
معماری یک GRN تأثیر زیادی بر ویژگیهای عملکردی آن دارد، از جمله پایداری، مقاومت، سازگاری و توانایی پردازش اطلاعات زیستی. درست مانند سیستمهای کنترلی مهندسیشده، شبکههای زیستی به صورت تصادفی ساخته نمیشوند؛ بلکه در طول تکامل، الگوهای خاصی از اتصال و موتیفهای شبکهای شکل گرفتهاند که مزیت انتخابی در شرایط فیزیولوژیک و محیطی موجودات فراهم میکنند. مطالعه معماری GRNها به ما کمک میکند تا فاصله بین مکانیزمهای مولکولی و رفتارهای سطح سیستم را پر کنیم.
۳.۱ توپولوژیهای شبکه
توپولوژی یک GRN الگوی کلی ارتباطات بین اجزای آن را توصیف میکند.
-
در سادهترین حالت، یک شبکه میتواند خطی باشد، یعنی یک زنجیره پیدرپی از رویدادهای تنظیمی که در آن یک ژن، ژن بعدی را به صورت یکطرفه فعال میکند.
-
اما در اغلب سیستمهای زیستی، توپولوژی غیرخطی است و شامل شاخهها، حلقههای بازخوردی (Feedback Loops)، ساختارهای پیشخور (Feed-forward) و تعاملات متقاطع بین مسیرهای مختلف میشود.
این آرایشهای پیچیده برای ایجاد رفتارهایی مانند:
دوپایداری (Bistability)، نوسانها (Oscillations)، کاهش نویز (Noise buffering) و بیان شرطی ژنها ضروری هستند.
یک ویژگی رایج بسیاری از شبکههای زیستی، توپولوژی مقیاس-آزاد (Scale-free) است؛ یعنی بیشتر گرهها تعداد کمی ارتباط دارند، در حالی که تعداد کمی از گرهها (هابها) تعداد بسیار زیادی اتصال دارند.
-
هابها معمولاً همان تنظیمکنندههای اصلی (Master Regulators) هستند، مثل فاکتورهای رونویسی یا پروتئینهای سیگنالدهنده که مجموعه بزرگی از ژنها را کنترل میکنند.
-
این ساختار مقاومت شبکه در برابر تغییرات تصادفی را افزایش میدهد، اما در عین حال باعث آسیبپذیری در برابر حمله هدفمند به هابها میشود.
ویژگی دیگر ماژولار بودن (Modularity) است؛ یعنی شبکه به زیرشبکهها یا ماژولها تقسیم میشود که هر کدام عملکرد نیمهمستقل دارند.
مثلاً:
-
یک ماژول میتواند ژنهای مسیر رشد را کنترل کند.
-
ماژول دیگر پاسخ به استرس را هدایت کند.
ماژولار بودن باعث میشود:
-
تکامل بتواند روی بخشی از شبکه اثر بگذارد بدون اینکه سایر بخشها را مختل کند.
-
کنترل ترکیبی (Combinatorial Control) امکانپذیر شود، جایی که ماژولها میتوانند دوباره ترکیب شوند تا عملکردهای جدید ایجاد کنند.
۳.۲ موتیفهای شبکه
در مقیاس کوچکتر، GRNها الگوهای تکرارشونده اتصال به نام موتیفهای شبکهای (Network Motifs) دارند که مانند مدارهای الکتریکی عمل میکنند.
نمونهها:
-
حلقه بازخورد منفی (Negative Feedback Loop): فاکتور رونویسی یک ژن را فعال میکند و آن ژن به نوبه خود همان فاکتور را سرکوب میکند.
-
مزیت: حفظ تعادل (Homeostasis) و کاهش نویز.
-
-
حلقه بازخورد مثبت (Positive Feedback Loop): یک تنظیمکننده، بیان خودش را (مستقیم یا غیرمستقیم) فعال میکند.
-
مزیت: ایجاد دوپایداری و حالتهای پایدار طولانیمدت (مثل تمایز سلولی).
-
-
حلقه پیشخور (Feed-forward Loop - FFL): تنظیمکننده A روی تنظیمکننده B اثر میگذارد و هر دو روی ژن C تأثیر میگذارند.
-
کارکردها: شناسایی سیگنالهای پایدار، تسریع پاسخ یا فیلتر کردن نویزهای گذرا.
-
همچنین موتیفهایی مانند:
-
سوئیچهای دوپایدار (Bistable Switches)
-
نوسانگرها (Oscillators) — مثل ریتمهای شبانهروزی که توسط حلقههای بازخورد با تأخیر زمانی ایجاد میشوند.
۳.۳ سازماندهی سلسلهمراتبی
بسیاری از GRNها ساختار سلسلهمراتبی (Hierarchical) دارند:
-
بالا: تنظیمکنندههای سراسری (Global Regulators) که گروههای بزرگی از ژنها را کنترل میکنند.
-
میانه: تنظیمکنندههای واسطه که در ماژولهای خاص فعالیت دارند.
-
پایین: تنظیمکنندههای نهایی که ژنهای ساختاری یا پروتئینهای اجرایی را کنترل میکنند.
این ساختار:
-
هماهنگی بین تصمیمگیری مرکزی و اجرای محلی را ممکن میکند.
-
در نمو جنینی، ژنهای اثر مادری در بالا قرار میگیرند و مسیرهای منطقهای و تمایزی را فعال میکنند.
۳.۴ گفتوگو و همبندی (Cross-talk & Interconnectivity)
گرچه سلسلهمراتب نظم ایجاد میکند، GRNها به صورت درختی خالص نیستند. تعاملات متقاطع بین ماژولها باعث یکپارچهسازی مسیرهای مختلف میشود.
مثلاً:
-
در گیاهان، شبکه کنترل زمان گلدهی ورودیهایی از نور، دما و هورمونها را ترکیب میکند.
-
در سلولهای ایمنی، مسیرهای شناسایی پاتوژن با مسیرهای سیگنالدهی سیتوکین تعامل دارند تا پاسخ دفاعی متعادل ایجاد شود.
این اتصالها باعث:
-
افزایش پایداری با وجود تنظیمکنندههای چندگانه برای یک ژن (Redundancy)
-
منطق AND/OR ژنتیکی (وقتی بیان ژن تنها با چند شرط همزمان فعال شود).
۳.۵ پلاستیسیته شبکه
پلاستیسیته (Plasticity) یعنی توانایی تغییر اتصالات و توپولوژی شبکه در اثر جهشها، تغییرات محیطی یا سیگنالهای تکوینی.
-
این ویژگی باعث سازگاری موجودات با شرایط جدید و نقش کلیدی در تکامل دارد.
-
مثال: تکثیر ژن میتواند نسخه اضافی یک تنظیمکننده ایجاد کند که بعداً عملکرد جدید پیدا کند.
در بیماریها (مثل سرطان)، پلاستیسیته به شکل منفی عمل میکند:
-
جهشها و تغییرات اپیژنتیک شبکه را بازسیمکشی میکنند تا رشد کنترلنشده، مقاومت به مرگ سلولی و فرار از سیستم ایمنی ایجاد شود.
۴. پویاییهای شبکههای تنظیم ژن (GRN Dynamics)
در حالی که ساختار یک شبکه تنظیم ژن (GRN) الگوها و ظرفیتهای بالقوه تنظیم را تعیین میکند، پویاییهای آن توصیف میکنند که وضعیت شبکه در طول زمان و در پاسخ به سیگنالهای داخلی و خارجی چگونه تغییر میکند. پویاییها بُعد زمانی بیان ژن را شامل میشوند، از جمله سرعت واکنشها، پایداری حالات، قابلیت نوسان و توانایی پردازش ورودیهای متغیر یا نویزی. درک کامل پویاییهای GRN برای پیشبینی رفتار سلولها در شرایط فیزیولوژیک، نحوه سازگاری آنها با تغییرات محیطی، و چگونگی بروز بیماریها در اثر اختلالات، ضروری است. این بخش به بررسی چارچوبهای نظری، مشاهدات تجربی و رویکردهای محاسباتی برای مطالعه رفتار پویا در GRN میپردازد.
۴.۱ فضای حالت و چشمانداز بیان ژن
یک روش قدرتمند برای درک پویایی GRN استفاده از مدل فضای حالت است که در آن هر ترکیب ممکن از سطوح بیان ژن به عنوان یک نقطه در فضایی چندبُعدی نمایش داده میشود. حرکت سیستم در این فضا در طول زمان نشاندهنده تغییر الگوهای بیان ژن در پاسخ به محرکها یا طی گذارهای تکوینی است. الگوهای بیان پایدار متناظر با حالتهای جاذب (Attractor States) هستند که میتوان آنها را به شکل درههایی در یک «چشمانداز بیان ژن» مجازی تجسم کرد. مدل چشمانداز اپیژنتیکی وادینگتون نمونه کلاسیکی از این ایده است، جایی که مسیر تمایز یک سلول مانند توپکی است که از شیبهای منشعب به سمت درههای خاص (حالتهای پایدار) حرکت میکند.
این حالتهای جاذب میتوانند نمایانگر انواع سلولی پایدار، حالات فعال گذرا یا شرایط پاتولوژیک باشند. عمق و شکل این درهها میزان پایداری یک حالت را تعیین میکند: درههای عمیقتر به حالتهایی با مقاومت بالاتر در برابر تغییر اشاره دارند، در حالی که درههای کمعمق، حالتهایی با برگشتپذیری آسانتر را نشان میدهند. تغییر بین جاذبها میتواند از طریق ورودیهای تنظیمی، نوسانات تصادفی یا مداخلات خارجی اتفاق بیفتد و فهم ویژگیهای شبکه که این تغییرات را کنترل میکنند، از مسائل اصلی زیستشناسی سامانههاست.
۴.۲ مدلهای ریاضی پویایی GRN
رفتار GRN را میتوان با استفاده از مدلهای ریاضی مختلف توصیف کرد که هر کدام مزایا و محدودیتهای خاص خود را دارند. معادلات دیفرانسیل معمولی (ODE) یکی از رایجترین روشهاست که تغییرات پیوسته سطح بیان ژن را با در نظر گرفتن نرخهای رونویسی، ترجمه و تخریب مولکولها مدل میکند. این مدلها میتوانند حالات پایدار و رفتارهای گذرا را شبیهسازی کرده و امکان تحلیل پایداری و نوسانی بودن سیستم را فراهم کنند. در مقابل، مدلهای شبکه بولی (Boolean Networks) ژنها را به صورت حالتهای گسسته روشن/خاموش در نظر میگیرند که اگرچه سادهترند، اما میتوانند منطق اصلی تنظیم را در شبکههای بزرگ بهخوبی نمایش دهند.
مدلهای پیشرفتهتر تصادفی (Stochastic) نیز وجود دارند که ماهیت ذاتاً تصادفی فرآیند رونویسی و ترجمه را در نظر میگیرند. معادلات دیفرانسیل تصادفی یا شبیهسازیهای مبتنی بر الگوریتم گیلِسپی قادر به بازتولید نوسانات مشاهدهشده در دادههای تکسلولی (مانند دادههای scRNA-seq) هستند. همچنین مدلهای ترکیبی که عناصر قطعی و تصادفی را با هم ترکیب میکنند، میتوانند هم رفتار میانگین جمعیت و هم تنوع بین سلولها را شبیهسازی کنند.
۴.۳ پایداری، دوپایداری و چندپایداری
یکی از پرسشهای بنیادین در پویایی GRN این است که آیا یک ساختار تنظیمی خاص منجر به یک حالت پایدار واحد میشود یا چند حالت پایدار. سامانههای تکپایدار (Monostable) به یک پروفایل بیان واحد همگرا میشوند و برای کارکردهایی که نیاز به ثبات بالا دارند مناسباند. سامانههای دوپایدار (Bistable) دارای دو جاذب مجزا هستند و رفتارشان شبیه یک کلید روشن/خاموش است. در این سیستمها، یک ورودی گذرا میتواند شبکه را بهطور دائمی از یک حالت به حالت دیگر منتقل کند، که این ویژگی در تعیین سرنوشت سلولی کاربرد زیادی دارد. سامانههای چندپایدار (Multistable) این قابلیت را به سه یا چند حالت پایدار گسترش میدهند و امکان وجود فنوتیپهای پایدار متنوع را در یک چارچوب ژنتیکی فراهم میکنند.
از دیدگاه ریاضی، تحلیل پایداری معمولاً با بررسی مقادیر ویژه (Eigenvalues) ماتریس ژاکوبین سیستم در نقاط تعادل انجام میشود. اگر بخش حقیقی این مقادیر منفی باشد، سیستم پایدار است؛ اگر مثبت باشد، سیستم ناپایدار بوده و احتمال تغییر حالت وجود دارد. از نظر زیستی، وجود چندپایداری امکان حافظهدار بودن سیستمهای سلولی را فراهم میکند، یعنی وقایع گذشته میتوانند بر رفتار فعلی تأثیرگذار باشند، حتی اگر محرک اولیه حذف شده باشد.
۴.۴ نوسانات و بیان ژن ریتمیک
برخی GRNها رفتار نوسانی نشان میدهند که در آن سطوح بیان ژن به صورت دورهای افزایش و کاهش مییابد. این نوسانات میتوانند ناشی از حلقههای بازخورد منفی با تأخیر ذاتی باشند، مانند شبکههای ساعت شبانهروزی (Circadian Clock)، تنظیم چرخه سلولی، یا ساعت سگمنتیشن در تکوین جنینی. نوسانات ممکن است تحت شرایط ثابت بهطور نامحدود ادامه پیدا کنند یا با گذر زمان کاهش یابند که این به قدرت و ساختار بازخورد بستگی دارد.
مدلهای نظری نشان میدهند که برای داشتن نوسانات پایدار، ترکیب بازخورد منفی و تأخیر زمانی مناسب ضروری است. این تأخیر میتواند از فرآیندهایی مانند فعالسازی رونویسی همراه با تجمع آهسته پروتئین یا مسیرهای سیگنالدهی چندمرحلهای ناشی شود. از نظر تجربی، نوسانات با استفاده از اندازهگیریهای سری زمانی mRNA یا پروتئین و اغلب با تصویربرداری زنده سلولی شناسایی میشوند.
۴.۵ نویز و اثرات تصادفی در پویایی GRN
بیان ژن ذاتاً نویزدار است، زیرا تعاملات مولکولی بهطور تصادفی رخ میدهند. حتی سلولهای ژنتیکی یکسان در شرایط یکسان، میتوانند سطوح بیان متفاوتی داشته باشند. این نویز میتواند درونی (Intrinsic) باشد که از رویدادهای تصادفی در رونویسی و ترجمه ناشی میشود، یا بیرونی (Extrinsic) که از نوسانات در اجزای سلولی مانند RNA پلیمراز، ریبوزوم یا متابولیتها ناشی میشود.
شبکههای تنظیم ژن میتوانند نویز را سرکوب کنند زمانی که دقت بیان اهمیت دارد، یا از نویز استفاده کنند برای اهدافی مانند تنوعبخشی و سازگاری در محیطهای متغیر. حلقههای بازخورد، حلقههای پیشخور و تکرار مسیرهای تنظیمی میتوانند نویز را کاهش دهند، در حالی که سامانههای چندپایدار میتوانند با کمک نویز به تغییر حالت خودانگیخته بین حالات مختلف دست پیدا کنند. کمیسازی نویز و مدلسازی آن نیازمند دادههای اندازهگیری تکسلولی و شبیهسازیهای تصادفی است که با پیشرفت فناوریهای تکسلولی، بسیار قدرتمندتر شدهاند.
۴.۶ ورودیهای خارجی و حسگری محیطی
پویاییهای GRN از محیط جدا نیستند؛ بلکه دائماً تحت تأثیر سیگنالهای خارجی مانند مواد غذایی، هورمونها، عوامل استرسزا و ارتباط سلول به سلول قرار دارند. این ورودیها میتوانند پارامترهای شبکه را تغییر دهند، حالتهای جاذب را جابهجا کنند، پایداری را تغییر دهند یا نوسانات ایجاد کنند. به عنوان مثال، در سیستمهای کوئوروم سنسینگ باکتریایی، تراکم جمعیت کنترل فعالسازی ژنهای خاص را در دست دارد و منجر به رفتار هماهنگ در کل جمعیت میشود. در موجودات پرسلولی، شبکههای سیگنالدهی هورمونی با GRNها تعامل دارند تا پاسخهای سیستمیک را در برابر تغییرات تکوینی یا محیطی هماهنگ کنند.
ادغام ورودیهای محیطی در پویایی GRN به سلولها اجازه میدهد که در شرایط متغیر سازگار شوند در حالی که ثبات داخلی خود را حفظ میکنند. این تعادل بین پایداری و انعطافپذیری یکی از ویژگیهای شاخص سیستمهای زیستی است و در زیستشناسی سنتتیک اهمیت زیادی دارد، جایی که باید GRNهای طراحیشده در محیطهای پیچیده و متغیر بهطور قابل اعتماد عمل کنند.
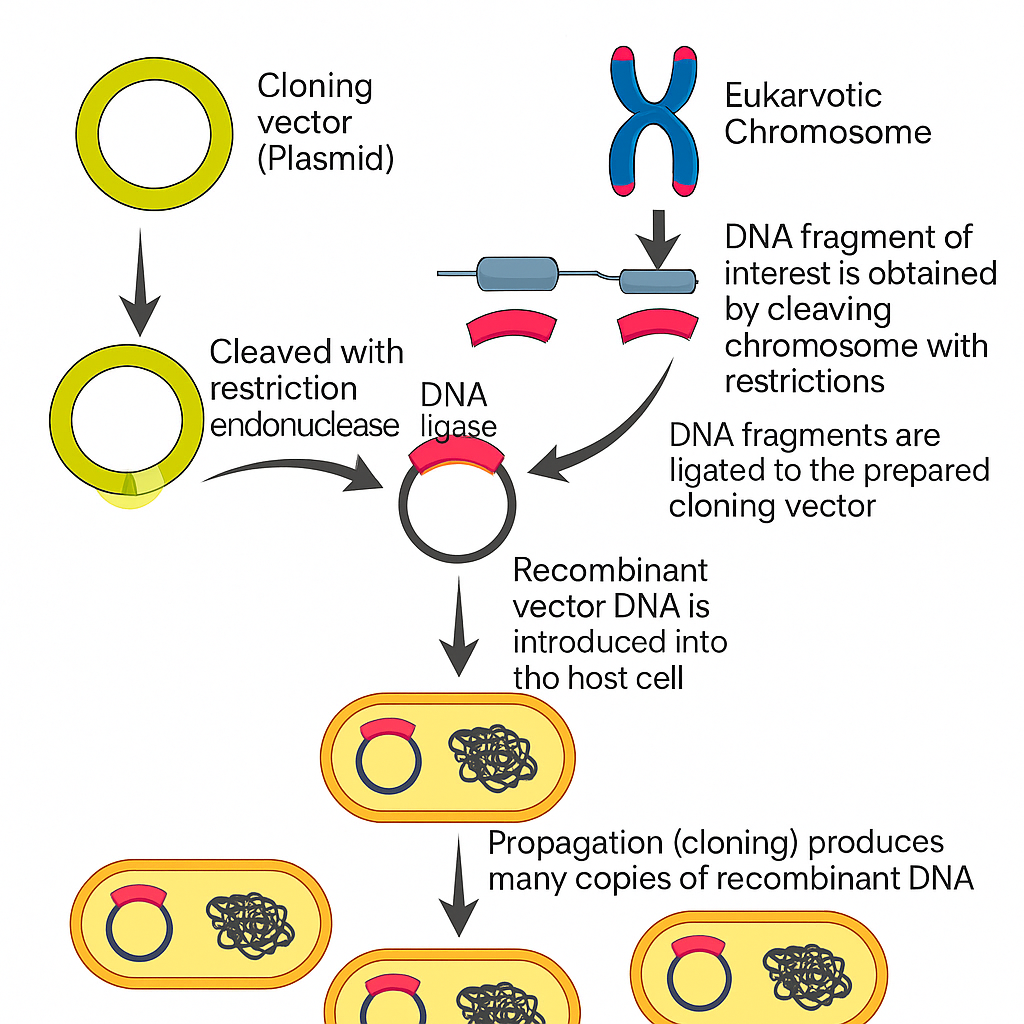
بخش ۵: روشهای بازسازی و تحلیل شبکههای تنظیم ژنی (GRNs)
بازسازی و تحلیل شبکههای تنظیم ژنی (GRNs) نیازمند ترکیبی از روشهای آزمایشی برای تولید داده و الگوریتمهای محاسباتی برای استنتاج روابط تنظیمی از آن دادههاست.
در دو دههی گذشته، ترکیب فناوریهای پروفایلگیری مولکولی با توان بالا و مدلسازیهای پیشرفتهی محاسباتی، پژوهش در حوزهی GRN را از نقشهبرداریهای کوچک و فرضیهمحور به سمت بازسازیهای در مقیاس ژنوم و دادهمحور سوق داده است.
روشهای مورد استفاده را میتوان به دو دستهی کلی تقسیم کرد:
-
روشهای آزمایشی → دادههای خام مولکولی را فراهم میکنند.
-
روشهای محاسباتی → ساختار شبکه را استنتاج، مدلسازی و تحلیل میکنند.
بازسازی مؤثر GRN به ترکیب هر دو دسته روش وابسته است، زیرا دادههای آزمایشی بهتنهایی معمولاً تمام ساختار شبکه را نشان نمیدهند، و پیشبینیهای محاسباتی نیز نیاز به تأیید آزمایشی دارند.
۵.۱ دیدگاه آزمایشی
-
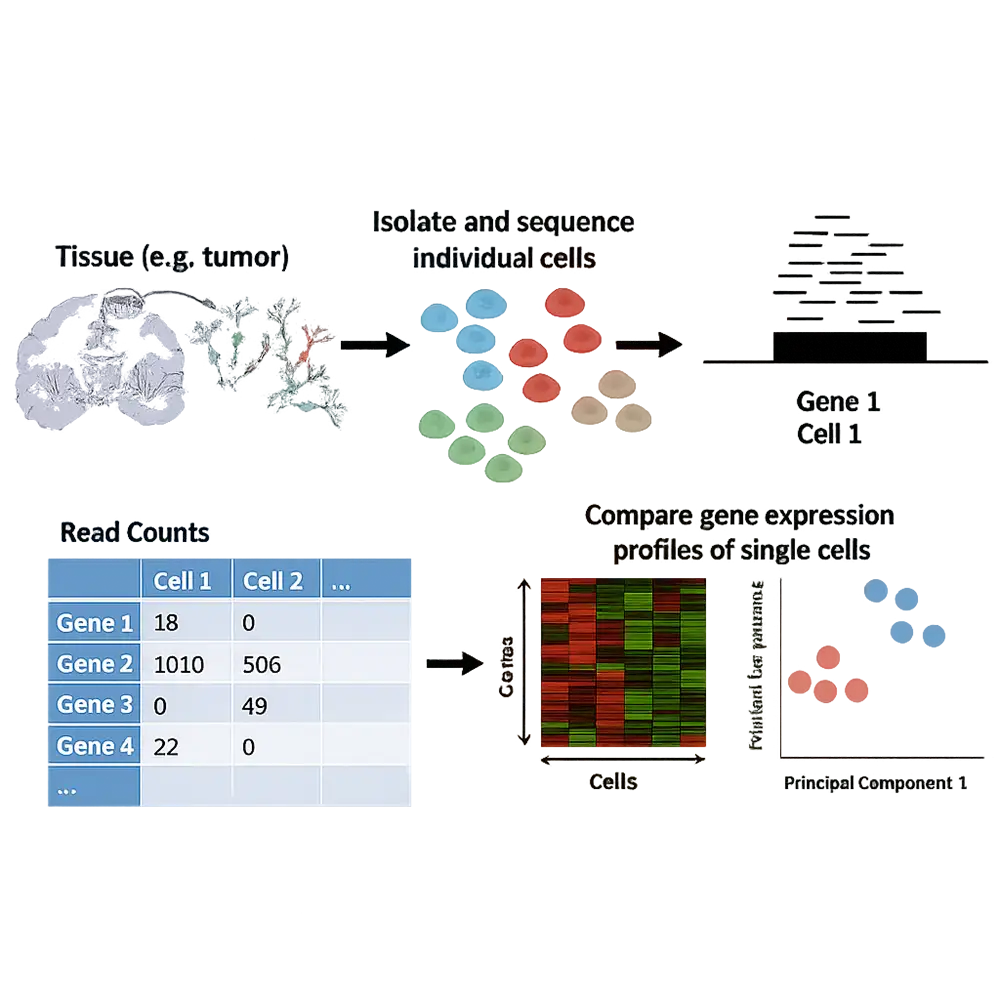
پروفایلگیری ترنسکریپتوم (Transcriptomic profiling) همچنان منبع اصلی داده برای استنتاج GRN محسوب میشود.
-
RNA-seq (در حالت کلی و تکسلولی) مقادیر mRNA را برای هزاران ژن بهطور همزمان اندازهگیری میکند.
-
RNA-seq تکسلولی (scRNA-seq) اهمیت ویژهای دارد چون تفاوتهای بین سلولها را آشکار کرده و برنامههای تنظیمی مرتبط با حالتها یا دودمانهای خاص سلولی را شناسایی میکند.
-
ChIP-seq و روشهای مشابه مانند CUT&RUN و CUT&Tag، شواهد مستقیمی از محلهای اتصال فاکتورهای رونویسی (TFs) روی ژنوم ارائه میدهند.
-
ATAC-seq، نواحی کروماتین باز را شناسایی میکند که معمولاً عناصر تنظیمی بالقوه هستند.
-
پلتفرمهای چند-اُمیک (Multi-omics) با ترکیب دادههای ترنسکریپتوم، اپیژنوم و پروتئوم در یک سلول یا نمونه، امکان پیوند تغییرات بیان ژن با دسترسی کروماتین و الگوهای اتصال TFها را فراهم میکنند.
۵.۲ دیدگاه محاسباتی
روشهای محاسباتی استنتاج GRN را میتوان به چند گروه اصلی تقسیم کرد:
-
روشهای همبستگیمحور → مانند WGCNA که ماژولهای ژنی همبیان شده را شناسایی میکنند.
-
روشهای مبتنی بر نظریه اطلاعات → مانند ARACNe که با استفاده از اطلاعات متقابل (Mutual Information) هم وابستگیهای خطی و هم غیرخطی بین پروفایلهای بیان ژن را تشخیص میدهند.
-
روشهای رگرسیونمحور → مانند GENIE3 و TIGRESS که بیان هر ژن را به عنوان تابعی از تنظیمکنندههای بالقوه مدلسازی کرده و با یادگیری ماشین (مثلاً جنگل تصادفی یا رگرسیون LASSO) آنها را رتبهبندی میکنند.
-
شبکههای بیزین → که بهصورت مدلهای گرافیکی احتمالاتی روابط تنظیمی را بهصورت یالهای جهتدار نمایش داده و حتی میتوانند پویایی زمانی را مدل کنند.
۵.۳ دادههای سری زمانی و روابط علّی
-
اضافه کردن دادههای سری زمانی پیشرفت بزرگی بوده، چون ترتیب رخدادهای تنظیمی را مشخص میکند.
-
روشهایی مانند شبکههای بیزین پویا (DBN) و مدلهای معادلات دیفرانسیل (ODE) از تغییرات زمانی بیان ژن برای استنتاج روابط علّی استفاده میکنند، نه صرفاً همبستگی.
۵.۴ دادههای حاصل از دستکاریهای ژنتیکی
-
دادههای برهمزننده (Perturbation-based) از طریق CRISPRi، CRISPRa، RNAi یا افزایش بیان TF به دست میآیند.
-
این روشها نتایج مستقیم تغییر یک تنظیمکننده را نشان میدهند و به عنوان واقعیت پایه (Ground Truth) برای ارزیابی پیشبینیهای محاسباتی استفاده میشوند.
-
Perturb-seq، ترکیب ترنسکریپتوم تکسلولی با دستکاری CRISPR در مقیاس گسترده است که بهطور سیستماتیک اثرات تنظیمی را در سطح ژنوم نقشهبرداری میکند.
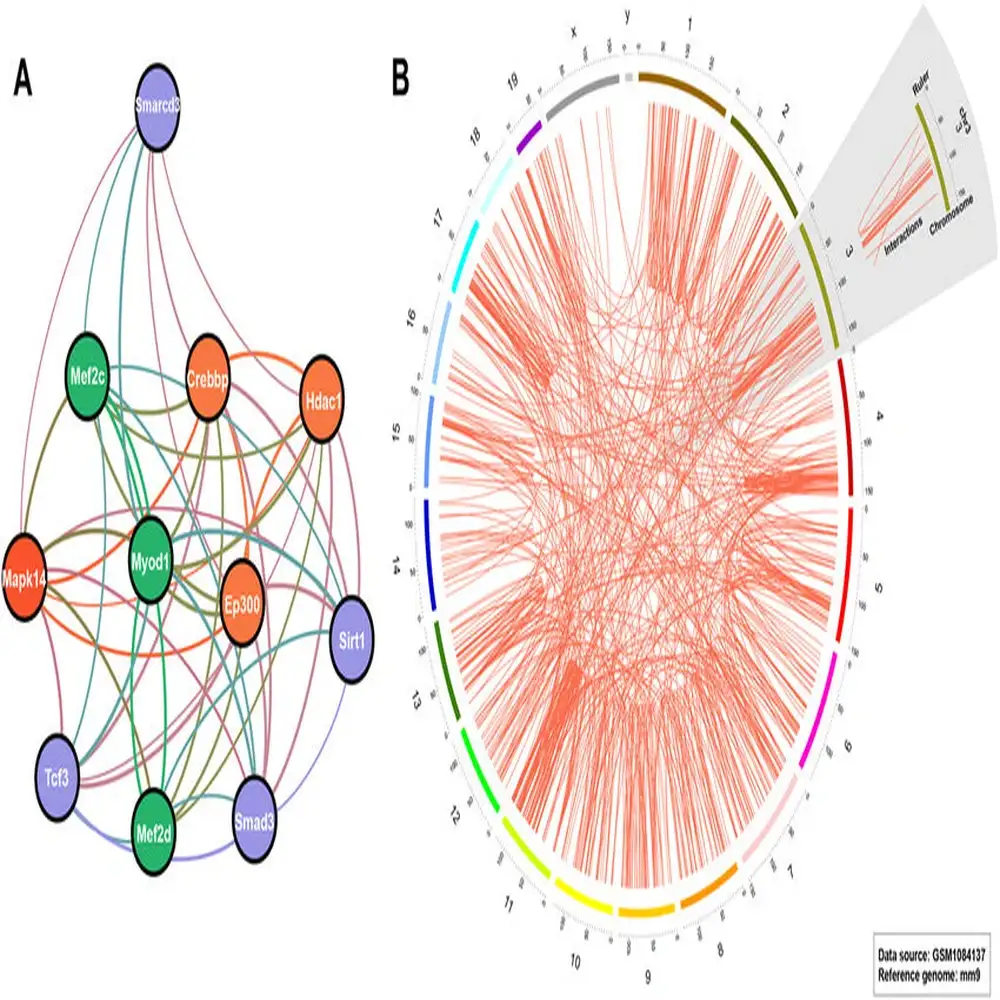
۵.۵ تحلیل ساختار شبکه
بعد از بازسازی GRN، ابزارهای نظریه گراف برای شناسایی ویژگیهایی مانند:
-
توزیع درجه اتصال (Degree distribution)
-
ضریب خوشهبندی (Clustering coefficient)
-
معیارهای مرکزیت (Centrality measures) → که تنظیمکنندههای کلیدی (Hub Regulators) و سازمان ماژولار را آشکار میکنند.
همچنین، حاشیهنویسی عملکردی (Functional annotation) با استفاده از:
-
پایگاههای مسیرهای زیستی
-
اصطلاحات Gene Ontology
-
شبکههای تعامل پروتئین–پروتئین
برای قرار دادن ماژولهای تنظیمی در زمینهی فرآیندهای زیستی شناختهشده انجام میشود.
۵.۶ روندهای نوین و یادگیری عمیق
-
چارچوبهای چند-مدلی (Multi-modal) با ترکیب دادههای مختلف مثل ترنسکریپتوم، اپیژنوم و دادههای ساختار سهبعدی کروماتین (Hi-C) شبکههایی دقیقتر و قابل تفسیرتر میسازند.
-
مدلهای یادگیری عمیق مانند شبکههای عصبی گرافی (GNN) و معماریهای مبتنی بر توجه (Attention-based) میتوانند وابستگیهای پیچیده و غیرخطی را شناسایی کنند.
-
این مدلها به دادههای بزرگ و دقیق و اعتبارسنجی محتاطانه نیاز دارند تا از بیشبرازش جلوگیری شود.
۵.۷ چرخه بازسازی و بهبود
بازسازی و تحلیل GRN یک فرآیند تکرارشونده است:
-
پیشبینیهای محاسباتی → آزمایش و اعتبارسنجی
-
دادههای جدید → بهبود مدلها
قویترین پژوهشها از چرخه بسته شامل تولید داده، مدلسازی، دستکاری آزمایشی و بازمدلسازی استفاده میکنند.
با پیشرفت توان تولید داده و پیچیدگی الگوریتمها، نقشهبرداری GRN در سطح تکسلول، در شرایط مختلف و با استنتاج علّی واقعی بهطور فزایندهای قابل دستیابی شده است.
بخش ۶: کاربردهای شبکههای تنظیم ژن (GRNs) در تحقیقات زیستپزشکی
شبکههای تنظیم ژن یا GRNها، بهعنوان چارچوبهای قدرتمندی برای درک برنامههای مولکولی پیچیده که عملکرد سلول را در سلامتی و بیماری کنترل میکنند، مطرح شدهاند. با ترسیم تعاملات بین فاکتورهای رونویسی، عناصر تنظیمی و ژنهای هدف، این شبکهها یک دیدگاه مکانیکی ارائه میدهند که به کمک آن میتوان مجموعههای بزرگ دادههای ژنومی و ترنسکریپتومی را تفسیر کرد. در تحقیقات زیستپزشکی، کاربردهای GRNها از کشفهای بنیادی بیولوژیکی گرفته تا نوآوریهای ترجمانی در تشخیص، درمان و پزشکی بازساختی گسترده است. این بخش به بررسی دقیق این کاربردها میپردازد و نشان میدهد که چگونه تحلیل GRN میتواند دادههای مولکولی را به بینشهای عملی زیستی تبدیل کند.
۱. شناسایی مکانیسمهای بیماری
یکی از مهمترین کاربردهای GRN، روشنکردن مکانیسم بیماریها است. بسیاری از بیماریها، از جمله سرطان، اختلالات نورودژنراتیو و بیماریهای خودایمنی، ناشی از اختلال در برنامههای تنظیمی هستند نه صرفاً جهش در نواحی کدکننده پروتئین. با مقایسه GRNهای بافت سالم و بیمار، پژوهشگران میتوانند تغییرات در معماری شبکه مانند ایجاد یا از دست دادن اتصالات تنظیمی یا ظهور تنظیمکنندههای مرکزی غیرطبیعی را شناسایی کنند که باعث پیشرفت بیماری میشوند.
برای مثال:
-
در سرطانشناسی، GRNهای خاص تومور میتوانند فاکتورهای رونویسی اصلی که برنامههای سرطانزا را هدایت میکنند، آشکار کنند.
-
در تحقیقات بیماریهای نورودژنراتیو، تحلیل GRN ماژولهای ژنی مرتبط با بقای نورون، عملکرد سیناپسی و پاسخهای التهابی را شناسایی کرده که اهداف بالقوهای برای درمان هستند.
۲. کشف نشانگرهای زیستی (Biomarkers)
GRNها در کشف بیومارکرها نیز نقش مهمی دارند. برخلاف نشانگرهای تکژنی، بیومارکرهای مبتنی بر شبکه فعالیت هماهنگ چندین ژن و تنظیمکننده را در بر میگیرند که اغلب قدرت پیشبینی و پایداری بیشتری در گروههای مختلف بیماران دارند.
-
برای مثال، امضاهای مشتقشده از GRN میتوانند پاسخ بیمار به ایمونوتراپی، حساسیت به شیمیدرمانی یا احتمال عود بیماری را پیشبینی کنند.
-
ترکیب دادههای GRN با چند-اُمیک (اپیژنومیکس، پروتئومیکس، متابولومیکس) بیومارکرهای چندلایهای ایجاد میکند که پیچیدگی واقعی سیستمهای زیستی را منعکس میکنند.
۳. شناسایی و بازاستفاده از اهداف دارویی
GRNها امکان شناسایی گرههایی را فراهم میکنند که تغییر فعالیت آنها میتواند عملکرد طبیعی شبکه را بازیابی کند.
-
روشهای محاسباتی میتوانند شبیهسازی مداخلات درون GRN را انجام دهند تا اثرات مهار یا فعالسازی یک تنظیمکننده خاص را پیشبینی کنند.
-
فارماکولوژی شبکهای با تطبیق داروهای موجود به GRNها، فرصتهایی برای بازاستفاده از داروها فراهم میکند. مثلاً دارویی که برای بیماری التهابی تأیید شده، ممکن است فعالیت یک ماژول شبکهای پاتولوژیک در بیماری دیگر را معکوس کند.
۴. پزشکی بازساختی و زیستشناسی سلولهای بنیادی
در پزشکی بازساختی و سلولهای بنیادی، GRNها برای درک و کنترل تصمیمات سرنوشت سلولی ضروری هستند. تمایز سلولهای بنیادی پرتوان به سلولهای تخصصی با تغییرات هماهنگ بیان ژن انجام میشود که تحت کنترل یک GRN مرکزی است.
-
با ترسیم این مدارهای تنظیمی، میتوان فاکتورهای رونویسی کلیدی را شناسایی کرد که دستکاری آنها میتواند تمایز بهسمت یک رده خاص یا حتی بازبرنامهریزی سلولهای تمایزیافته به حالت پرتوان را القا کند.
۵. تعاملات میزبان–پاتوژن
در بیماریهای عفونی، تحلیل GRN در میزبان و پاتوژن میتواند استراتژیهای تنظیمی هر طرف را آشکار کند.
-
مثلاً در عفونت ویروسی، GRN میزبان ممکن است به سمت فعالسازی سیستم ایمنی حرکت کند، در حالی که عوامل ویروسی ممکن است فاکتورهای رونویسی میزبان را برای سرکوب پاسخ ضدویروسی هدف قرار دهند.
۶. پزشکی دقیق (Precision Medicine)
پزشکی شخصی با کمک تحلیل GRN تقویت میشود، زیرا تغییرات ژنومی خاص هر بیمار در چارچوب یک شبکه تفسیر میشود. این رویکرد میتواند آسیبپذیریهای منحصربهفرد در زیستشناسی بیماری هر فرد را شناسایی کرده و استراتژیهای درمانی واقعاً شخصیسازیشده ارائه دهد. ترکیب تحلیل GRN با مدلهای یادگیری ماشین نیز میتواند پیشبینی پاسخ به درمان را بهبود بخشد.
جمعبندی
GRNها یک چارچوب یکپارچه برای تفسیر دادههای زیستی پیچیده و تبدیل آنها به بینشهای بالینی فراهم میکنند. کاربردهای آنها از شناسایی مکانیسم بیماریها تا هدایت توسعه درمانها و تشخیصهای نوین گسترده است. با پیشرفت روشهای تجربی و محاسباتی، GRNها بیش از پیش به ابزار کلیدی در تحقیقات زیستپزشکی تبدیل خواهند شد.
بخش ۷: چالشها و محدودیتهای پژوهش در زمینه شبکههای تنظیم ژن (GRN)
با وجود پتانسیل تحولآفرین شبکههای تنظیم ژن (GRNs) در درک سیستمهای زیستی و پیشبرد تحقیقات پزشکی، بازسازی و تفسیر این شبکهها با چالشهای زیادی مواجه است. این محدودیتها هم از موانع فنی در تولید داده و هم از پیچیدگیهای مفهومی در مدلسازی فرآیندهای زیستی ناشی میشوند. رفع این چالشها برای تولید شبکههایی دقیق، قابل بازتولید و زیستشناسیمحور ضروری است.
۱. دادههای ناقص و پر از نویز
یکی از مهمترین موانع، ناقص و نویزی بودن دادههای آزمایشگاهی است. فناوریهای پیشرفته مانند RNA-seq، توالییابی RNA تکسلولی (scRNA-seq) و آزمونهای دسترسی کروماتین حجم زیادی از داده تولید میکنند، اما:
-
در معرض تغییرات فنی، اثرات دستهای (Batch Effects) و حذف اتفاقی دادهها (Dropout) هستند، بهویژه در آزمایشهای تکسلولی.
-
این مشکلات میتوانند روابط واقعی زیستی را پنهان کرده و اتصالات اشتباه را وارد GRN کنند.
-
همچنین، دامنه دینامیکی و حساسیت این فناوریها ممکن است نتواند رونوشتهای کممقدار یا برهمکنشهای تنظیمی گذرا را شناسایی کند.
۲. دشواری در تشخیص علیت
بسیاری از روشهای محاسباتی بر همبستگی آماری یا اطلاعات متقابل بین پروفایل بیان ژن تکیه دارند.
-
این روشها ممکن است ارتباطهای بالقوه را پیشنهاد دهند، اما لزوماً تنظیم مستقیم یا علّی را نشان نمیدهند.
-
تمایز بین برهمکنش مستقیم فاکتور رونویسی–ژن هدف و اثرات غیرمستقیم (از طریق ژنهای واسطه) یک چالش اساسی است.
-
اعتبارسنجی آزمایشی (مانند دستکاری CRISPR یا ChIP-seq) ضروری است، اما بسیار هزینهبر و زمانبر است.
۳. وابستگی به بستر (Context-dependence)
برنامههای تنظیمی میتوانند بهشدت بین انواع سلول، مراحل رشد، شرایط محیطی یا وضعیت بیماری متفاوت باشند.
-
شبکهای که از یک بستر استخراج میشود ممکن است برای بستر دیگر نمایانگر دقیقی نباشد.
-
فاکتورهای رونویسی ممکن است چندعملکردی باشند: در یک موقعیت فعالکننده و در موقعیت دیگر سرکوبکننده، بسته به کو-فاکتورها و وضعیت کروماتین.
۴. محدودیتهای محاسباتی و عدم وجود استاندارد طلایی
-
استنتاج GRN، بهویژه با ادغام دادههای چند-اُمیک، بسیار منابعبر (Resource-intensive) است و برای دادههای تکسلولی با میلیونها سلول به قدرت پردازشی و حافظه بسیار زیاد نیاز دارد.
-
استاندارد طلایی جهانی برای ارزیابی دقت شبکههای بازسازیشده وجود ندارد.
-
منابع موجود (پایگاههای داده یا تعاملات استخراجشده از مقالات) ممکن است ناقص یا سوگیرانه به سمت ژنها و مسیرهای شناختهشده باشند.
۵. محدودیت وضوح زمانی و مکانی دادهها
-
اغلب مطالعات ترنسکریپتومیک، تنها تصویری لحظهای (Snapshot) از بیان ژن را ثبت میکنند.
-
این امر مدلسازی رفتار پویای GRNها در طول زمان را دشوار میکند.
-
فرآیندهای زیستی مانند رشد، پاسخ ایمنی یا سازگاری با استرس بهطور ذاتی دینامیک هستند و نیازمند دادههای سری زمانی یا مکانی دقیق میباشند.
۶. افزونگی زیستی و پایداری شبکه
-
بسیاری از GRNها دارای مسیرهای افزونه و حلقههای بازخورد هستند که پایداری سیستم را حفظ میکنند.
-
این افزونگی باعث میشود که دستکاری یک تنظیمکننده ممکن است اثر قابلتوجهی نداشته باشد، چون مسیرهای جبرانی فعال میشوند.
-
این موضوع اهمیت واقعی تعاملات را پنهان کرده و تفسیر آزمایشهای مداخلهای را دشوار میکند.
۷. چالش در ترجمه به کاربردهای بالینی
-
حرکت از پیشبینی محاسباتی به مداخلات بالینی تأییدشده نیازمند اعتبارسنجی گسترده، تأییدیههای قانونی و درنظر گرفتن تنوع بیماران است.
-
مسائل اخلاقی و لجستیکی مانند حریم خصوصی دادهها و استانداردسازی روشهای تحلیل نیز مانع ادغام GRNها در تصمیمگیری بالینی میشوند.
جمعبندی
اگرچه GRNها ابزار قدرتمندی برای روشنکردن پیچیدگی سیستمهای زیستی هستند، اما محققان باید با چالشهای فنی، مفهومی و عملی زیادی روبهرو شوند. پیشرفت در این حوزه وابسته به بهبود روشهای تجربی و الگوریتمهای محاسباتی و همچنین یکپارچگی بیشتر بین این دو است. با رفع این موانع، میتوان به سمت مدلهای دقیقتر، پویاتر و بستر-آگاهتر حرکت کرد که بازتابدهنده واقعیت پیچیده تنظیم ژن در موجودات زنده باشد.
بخش ۸: مسیرهای آینده در پژوهش شبکههای تنظیم ژن (GRN)
حوزه پژوهش شبکههای تنظیم ژن (GRNs) با سرعت زیادی در حال پیشرفت است و این رشد، حاصل پیشرفتهای فنی در روشهای آزمایشگاهی، مدلسازی محاسباتی و استراتژیهای یکپارچهسازی دادهها است. همانطور که پژوهشگران تلاش میکنند بر محدودیتهای بخش قبل غلبه کنند، چند مسیر نوظهور وجود دارد که میتواند بهطور قابلتوجهی دقت، وضوح و کاربردپذیری مدلهای GRN را بهبود دهد. این تحولات نه تنها درک ما از تنظیم ژن را عمیقتر میکند، بلکه فرآیند تبدیل یافتههای GRN به کاربردهای پزشکی عملی را نیز سرعت میبخشد.
۱. ادغام دادههای چند-اُمیک (Multi-omics Integration)
-
یکی از مسیرهای مهم، ادغام مجموعه دادههای چند-اُمیک برای ساخت GRNهای جامعتر و دقیقتر است.
-
ترکیب ترنسکریپتومیک، اپیژنومیک، پروتئومیک، متابولومیک و ساختار سهبعدی کروماتین به محققان امکان میدهد چندین لایه از تنظیم بیان ژن را شناسایی کنند.
-
این ادغام میتواند:
-
روابط مستقیم فاکتور رونویسی–ژن هدف را از اثرات غیرمستقیم جدا کند.
-
خطاهای مثبت کاذب را کاهش دهد.
-
مدلسازی تنظیم بستر-ویژه را ممکن سازد.
-
-
پلتفرمهای چند-اُمیک تکسلولی که همزمان بیان RNA، دسترسی کروماتین، متیلاسیون DNA و مقدار پروتئین را در یک سلول اندازهگیری میکنند، تحولی بزرگ در بازسازی شبکههای ویژه هر نوع سلول ایجاد خواهند کرد.
۲. ترنسکریپتومیک زمانی و مکانی با وضوح بالا
-
فناوریهایی مانند SLAM-seq، تحلیل سرعت RNA (RNA velocity) و تصویربرداری زنده سلول میتوانند تغییرات بیان ژن را در طول زمان دنبال کنند و ماهیت پویای GRNها را آشکار سازند.
-
ترنسکریپتومیک فضایی و روشهایی مثل هیبریداسیون درجا چندگانه (Multiplexed in situ hybridization) امکان نقشهبرداری بیان ژن در بافتهای دستنخورده را فراهم میکند.
-
ترکیب دادههای زمانی و مکانی به بازسازی GRNهایی منجر میشود که هم زمان و هم مکان برهمکنشهای تنظیمی را لحاظ میکنند؛ این کار نمایی واقعیتر از فرآیندهایی مانند رشد، بازسازی و پیشرفت بیماری ارائه میدهد.
۳. مدلهای محاسباتی و یادگیری ماشین پیشرفته
-
مدلهای یادگیری عمیق، شبکههای عصبی گرافی، و مدلهای گرافیکی احتمالاتی قادرند روابط غیرخطی و وابستگیهای پیچیده را در دادههای زیستی با ابعاد بالا شناسایی کنند.
-
استفاده از یادگیری انتقالی (Transfer learning) امکان بهرهگیری از دادههای مرتبط برای بهبود پیشبینی در سیستمهای کمتر مطالعهشده را فراهم میکند.
-
توسعه هوش مصنوعی قابل تفسیر (Explainable AI) تضمین میکند که پیشبینیهای مدلهای پیچیده قابل ردیابی به شواهد زیستی باشند.
۴. روشهای آزمایشگاهی مبتنی بر مداخله (Perturbation-based Methods)
-
غربالگریهای مبتنی بر CRISPR همراه با خوانش ترنسکریپتومیک، امکان آزمون سیستماتیک روابط تنظیمی پیشبینیشده را فراهم میکند.
-
روشهایی مانند Perturb-seq و CRISPRi/a در ترکیب با توالییابی تکسلولی، دستکاری دقیق فاکتورهای رونویسی و مشاهده اثرات پاییندستی را ممکن میسازند.
-
این روشها نه تنها اعتبار شبکهها را تأیید میکنند بلکه سازوکارهای علّی و سلسلهمراتب تنظیمی را نیز آشکار میسازند.
۵. ادغام GRNها در مدلهای پیشبینی بیماری
-
با نقشهبرداری از تعاملات تنظیمی مختلشده در بیماریها، میتوان نشانگرهای زیستی جدید، اهداف درمانی تازه و استراتژیهای دستهبندی بیماران را شناسایی کرد.
-
در سرطانشناسی، GRNها میتوانند برنامههای رونویسی محرک پیشرفت تومور، متاستاز یا مقاومت دارویی را آشکار کنند.
-
با واردکردن دادههای بیمار به مدلهای GRN، میتوان به سمت پزشکی شخصی حرکت کرد که درمانها بر اساس معماری تنظیمی منحصر به فرد بیماری هر فرد طراحی شوند.
۶. کاربردهای زیستفناوری سنتزی (Synthetic Biology)
-
طراحی مدارهای تنظیمی مصنوعی به محققان امکان میدهد سلولهایی با رفتار دلخواه (مثل تمایز کنترلشده، بهینهسازی متابولیسم، یا انتقال هدفمند درمان) ایجاد کنند.
-
پیشرفت در ویرایش ژنوم، پروموترهای سنتزی و اجزای تنظیمی ماژولار ساخت و آزمون GRNهای مصنوعی را ممکن میسازد.
-
این کاربردها GRN را از زیستشناسی توصیفی به مهندسی و درمان گسترش میدهند.
۷. استانداردسازی و اشتراک دادهها و ابزارها
-
ایجاد پایگاههای داده باز و سازگار از تعاملات تأییدشده آزمایشگاهی و همچنین خط لولههای محاسباتی استاندارد، بازتولیدپذیری و مقایسهپذیری مطالعات را تسهیل میکند.
-
همکاریهای بینالمللی که منابع و تخصصها را به اشتراک میگذارند، برای مقابله با پیچیدگی پروژههای GRN بزرگ ضروری خواهند بود.
جمعبندی
آینده پژوهش GRN با نوآوریهای فناورانه، پیشرفتهای محاسباتی و همکاریهای میانرشتهای شکل خواهد گرفت. با ادغام دادههای چند-اُمیک، پروفایلبرداری زمانی و مکانی دقیق، مدلسازی پیشرفته و اعتبارسنجی سختگیرانه آزمایشگاهی، این حوزه به سمت شبکههایی دقیقتر، پیشبینیپذیرتر و مرتبطتر با بالین حرکت خواهد کرد. این پیشرفتها میتوانند نه تنها فهم ما از تنظیم ژن را تعمیق بخشند، بلکه فرصتهای تازهای برای پزشکی دقیق، زیستفناوری سنتزی و مداخلات سیستمی در سلامت و بیماری فراهم کنند.